
2022年7月,清华大学统计学研究中心侯琳副教授课题组与生命科学学院张强锋副教授课题组合作在 Nature Communications 期刊上在线发表了题为“Differential analysis of RNA structure probing experiments at nucleotide resolution: uncovering regulatory functions of RNA structure ”的研究论文(https://www.nature.com/articles/s41467-022-31875-3)。该研究提出了一个兼容多种RNA结构探测数据的标准化及结构差异分析的计算框架。该方法可以在单碱基分辨率水平评估RNA结构差异区域,为探索RNA结构在不同生物学过程中发挥的作用提供强有力的支持。同时,该研究还开发了相应的软件包 DiffScan(https://github.com/yub18/DiffScan)供广大研究人员使用。清华大学统计学研究中心2018级博士研究生余博是该文章的第一作者,清华大学生命科学学院的博士毕业生李盼亦参与了该项研究。此外,该研究得到了国家自然科学基金和国家重点研发计划的支持。
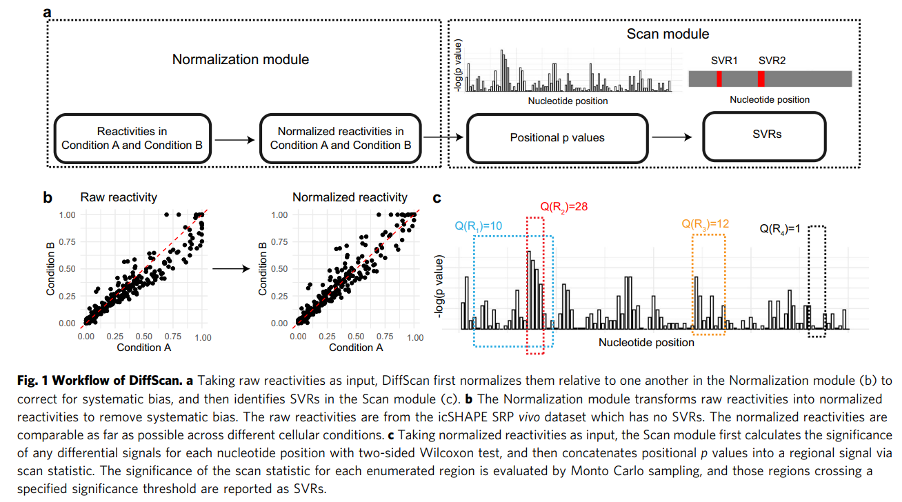
RNA通过形成特定的结构发挥相应的功能,并且RNA结构在不同的细胞环境中往往存在差异。因此,通过对不同条件下的RNA结构组数据进行准确的结构差异区域分析,对于揭示RNA在不同生物学过程中的功能及作用机制至关重要。考虑到高通量的RNA结构探索技术产生的实验数据往往存在高维度、高噪音等特点,RNA结构的差异分析存在较大难度。在本研究中,研究团队提出针对高通量结构探测数据的标准化和差异分析计算框架DiffScan,该方法首先对结构探测数据进行标准化以移除系统性偏差,再运用扫描统计量在RNA结构组中识别结构差异区域。
DiffScan 的独特贡献在于:(1)其标准化模块能有效地移除实验间存在的系统性偏差,并充分保留结构差异信号;(2)其扫描统计量能够在单碱基分辨率水平自适应地识别结构差异区域的位置与长度;(3)该计算框架可以兼容包括icSHAPE、DMS-seq在内的多种高通量RNA结构探测技术产生的实验数据。
研究团队将DiffScan应用于不同亚细胞的RNA结构组数据中,通过结合相关的基序富集分析阐明了RNA结构变化与mRNA丰度之间的潜在联系可能是由包括serine/arginine rich splicing factors在内的特定RNA结合蛋白所介导的。因此,该研究也证明了DiffScan是破译RNA结构组数据的有效手段。
